-1 Centro di Riferimento Regionale per le Emergenze Microbiologiche, Unità Operativa di Microbiologia, Azienda Ospedaliera-Universitaria Policlinico S.Orsola-Malpighi, Bologna;
Introduzione.
Le infezioni a carico dell'apparato respiratorio costituiscono ancora oggi una delle principali cause di malattia nella popolazione infantile. In particolare, le polmoniti ad eziologia infettiva mantengono un ruolo epidemiologico molto consistente nell'ambito della patologia respiratoria del paziente pediatrico (Sinaniotis et al., Current Opinion in Pulmonary Medicine 11: 218-225; 2005) . In quest'ambito, ai patogeni "classici", il cui rappresentante più importante, sia per epidemiologia sia per rilevanza clinica, resta Streptococcus pneumoniae, si sono recentemente aggiunti patogeni cosiddetti "atipici", fra cui la maggiore rilevanza epidemiologica va attribuita a Chlamydia pneumoniae e Mycoplasma pneumoniae (Esposito S. et al., European Respiratory Journal 17: 241-245; 2001). In quest'articolo saranno descritte la caratteristica dei maggiori patogeni respiratori dell'età infantile con particolare riguardo agli aspetti patogenetici e diagnostici di ciascun agente microbico, ponendo speciale attenzione ai nuovi metodi ora disponibili per eseguire la diagnosi di laboratorio delle infezioni da ciascuno di essi provocate.
Questo batterio Gram positivo resta una delle principali cause di polmone di comunità, sia nel-l'età adulta sia fra la popolazione pediatrica, in tutte le aree geografiche. Alcuni gruppi di popolazione sono esposti ad un maggiore rischio di contrarre infezioni ad eziologia pneumococcica: i bambini piccoli, gli anziani ed i soggetti immunocompromessi. Si stima che ogni anno, nel mondo, muoiano in seguito a gravi infezioni invasive, inclusa la polmonite, sostenute da pneumococco circa un milione di bambini d'età inferiore ai 5 anni. La principale difesa dell'organismo nei confronti delle infezioni da pneumococco è costituita dalla fagocitosi opsonica dipendente dalla risposta anticorpale specifica e dal complemento. Il meccanismo fondamentale che sta alla base di tutte le infezioni da S.pneumoniae è quello della colonizzazione delle cavità nasofaringee, che resta spesso un fatto totalmente silente dal punto di vista clinico, pur essendo il presupposto fondamentale per lo sviluppo di successive patologie pneumococciche a livello polmonare o sistemico. Nel bambino, la flora batterica che può colonizzare il nasofaringe comprende svariate specie microbiche, fra cui S.pneumoniae, Moraxella catarrhalis ed Haemophilus influenzae. La percentuale di soggetti colonizzati da questi microrganismi dipende dall'età, dall'area geografica e dal background genetico e socio-economico delle varie popolazioni: la colonizzazione nasofaringea è un processo dinamico fortemente condizionato da processi di competizione interspecifica, per cui la presenza di streptococchi a-emolitici nella flora nasofaringea può inibire la capacità di colonizzazione da parte di specie patogene quali S.pneumoniae. La capacità di S.pneumoniae di colonizzare l'epitelio respiratorio si base su legami che si stabiliscono fra recettori di natura saccaridica (N-acetil-glucosamina) presenti sulle membrane delle cellule epiteliali e specifiche adesine batteriche (ad esempio la cosiddetta pneumococcal surface adhesin A - PsaA). Il passaggio dalla colonizzazione asintomatica alla patologia invasiva richiede la produzione locale di mediatori flogistici (come interleuchina 1 e tumor necrosis factor a) che modificano il tipo ed il numero dei recettori disponibili sulle cellule epiteliali, consentendo ai batteri di sfruttare queste modificazioni per penetrare nelle cellule e, conseguentemente, invadere il circolo dando luogo alla diffusione batteriemica che è alla base delle patologie invasive da pneumococco. La diagnosi delle infezioni polmonari da pneumococco si basa classicamente sull'isolamento del patogeno dall'espettorato o da altri materiali biologici di provenienza polmonare profonda ottenuti con tecniche di prelievo invasive, quali lavaggio ed aspirazione bronchiale. Vista la presenza di una fase batteriemica nel quadro clinico precoce della polmonite da pneumococco, l'isolamento del patogeno è in alcuni casi ottenibile anche attraverso l'impiego dell'emocoltura. Recentemente sono stati descritti e valicati dal punto di vista clinico nuovi metodi per la diagnosi d'infezione da S.pneumoniae, fra cui vanno ricordati la ricerca dell'antigene urinario del pneumococco e svariati metodi basati sulla tecnologia d'amplificazione degli acidi nucleici (Nucleic Acid Amplification Test - NAATs) con tecnica do polymerase chain reaction (PCR). I metodi per la determinazione dell'antigene urinario di pneumococco si basano sulla tecnologia immunocromatografica e sono dotati di notevole specificità e sensibilità: questi test mantengono un certo livello di positività per tempi variabili anche dopo il trattamento antibiotico e la risoluzione clinica dell'infezione e, pertanto, sono da impiegarsi prevalentemente nel sospetto diagnostico di fase acuta di infezione. La tecnica di PCR per la diagnosi d'infezione da pneumococco è stata valutata con impiego su campioni ematici (sangue intero, plasma o siero) con sensibilità variabile dal 57% al 100%. L'impiego della tecnica PCR su campioni respira-tori ha dato esito a valori di sensibilità variabile fra il 68% ed il 100%. I maggiori dubbi relativamente all'impiego di tecniche NAATs per la diagnosi d'infezione da pneumococco nascono dalla possibilità di risultati falsamente positivi causati dalla erronea identificazione, per scarsa specificità dei metodi, di altri batteri colonizzanti il nasofaringe: da qui la necessità di impiegare target genetici di amplificazione specie specifici per S.pneumoniae. Fra i geni di pneumococco maggiormente usati come bersaglio delle tecniche PCR va ricordato quello della pneumolisina, la cui sequenza nucleotidica è stata considerata a lungo altamente specie-specifica. Recenti studi hanno messo in dubbio la specificità di questa sequenza genica in quanto geni con alto grado di similitudine sono stati identificati nel genoma d'altri streptococchi, quali ad esempio, il gene dell'autolisina di S.mitis. Al momento attuale non sono quindi disponibili metodi NAATs per la diagnosi d'infezione da S.pneumoniae che si possano impiegare nella routine dei laboratori di microbiologia: le tecniche PCR vanno pertanto considerate degli utili strumenti di ricerca in attesa di ricevere adeguate conferme di validazione clinica (Bogaert et al., The Lancet Infectious Diseases 4: 144 - 154; 2004).
Chlamydia pneumoniae.
Mycoplasma pneumoniae.
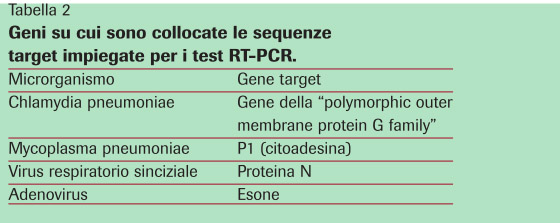
I micoplasmi rappresentano le più piccole forme biologiche in grado di replicarsi autonomamente, avendo dimensioni di 1-2 mm di lunghezza per 0.1-0.2 mm di diametro ed un genoma costituito da circa 820.000 basi con 687 geni identificati. Il ridotto genoma di M.pneumoniae e le sue limitate capacità biosintetiche per proteine, carboidrati e lipidi rendono ragione delle complesse necessità nutrizionali che devono essere soddisfatte per ottenere la crescita in vitro di questi batteri privi di parete cellulare. L'incidenza dell'eziologia da M.pneumoniae nell'ambito delle polmoniti di comunità si stima pari al 10-30% dei casi: queste forme polmonari rappresentano verosimilmente solo il 10% del totale delle infezioni respiratorie da micoplasma, essendo le forme prevalenti quelle delle alte vie e la tracheobronchite, mentre circa il 15% delle infezioni respiratorie possono restano sostanzialmente asintomatiche. Come sopra ricordato a proposito di C.pneumoniae, anche le polmoniti da M.pneumoniae sono tipicamente descritte come forme interstiziali o "atipiche" per distinguerle dalla classica forma da pneumococco. La polmonite da micoplasma è forma prevalente nel bambino e nel giovane adulto mentre la si osserva più raramente nell'anziano. La patogenicità di M.pneumoniae risiede principalmente nella capacità di aderire alle cellule dell'epitelio respiratorio e di penetrare all'interno delle cellule stesse mediante un processo di fusione delle membrane, cui consegue una possibile evoluzione in senso citolitico con induzione di una risposta flogistica: viene attualmente ipotizzato che la flogosi sia uno dei meccanismi principali alla base della sintomatologia respiratoria delle infezioni da micoplasmi. L'isolamento colturale di M.pneumoniae da materiali respiratori per ottenere la diagnosi di laboratorio di polmonite è possibile, ma resta un procedimento molto lungo e laborioso nonché costoso: la sensibilità di questa tecnica è di circa il 60% pur essendo la specificità del 100%, come ovvio (Waites et al., Clinical Microbiology Review 17: 697 - 728; 2004). Poiché le tecniche colturali sono scarsamente sensibili e di lunga esecuzione, la diagnosi d'infezione respiratoria da micoplasmi si ottiene frequentemente con l'impiego di tecniche sierologiche che soffrono di tutte le limitazioni sopra ricordate a proposito della diagnosi d'infezione da C.pneumoniae. Pertanto, anche nel caso delle infezioni da micoplasmi le tecniche NAATs sono state oggetto di un consistente numero di studi sin dalla fine degli anni 80 del secolo scorso. La diagnosi molecolare è stata valutata su differenti campioni clinici: escreato, lavaggio e aspirato bronchiale, secreto nasofaringeo aspirato nasofaringeo e fluido pleu-rico.Allo stesso modo sono stati valutati diversi metodi d'estrazione dell'acido nucleico dai campioni biologici ed è stata valutata l'efficienza di molte sequenze geniche da impiegare come bersaglio della reazione d'amplificazione: i geni maggiormente utilizzati come bersaglio delle reazioni di PCR sono stati il gene dell'operone dell'ATPasi, il gene P1 e il gene dello RNA ribosomiale 16S (16S rRNA) spesso con scelta di sequenze (e quindi prodotti di amplificazione) differenti posti sul medesimo gene. Anche i protocolli d'amplificazione sono stati svariati: dalla classica PCR, alla
PCR nested e multiplex (per la simultanea determinazione di più microrganismi in un solo ciclo d'amplificazione su di unico campioni patologico), fino alla Real Time PCR. Allo stato attuale delle conoscenze, il metodo che ha dimostrato la migliore performance diagnostica per le infezioni respiratorie da micoplasmi in età pediatrica si è dimostrata la Real Time PCR (che come sopra ricordato consente di ottenere risultati quantitativi,che meglio correlano con il quadro clinico dei piccoli pazienti) eseguita su prelievi di secreto faringeo (Koens et al., Journal of Clinical Microbiology 41: 4915 - 4923; 2003).
Altri batteri patogeni (Haemophilus influenzae, Moraxella catarrhalis, Legionella pneumophila).
Al momento esistono poche evidenze sperimenta-li relativamente all'impiego delle metodiche NAATs per la diagnosi delle infezioni respiratorie sostenute da H.influenzae e M.catarrhalis e pertanto la diagnosi di queste forme resta sostanzialmente basata sulle classiche metodologie d'isolamento colturale. In uno studio recente è stato dimostrato che la tecnica di PCR è sensibile e specifica quando applicata alla diagnosi di infezione polmonare da H.influenzae partendo da campioni ematici nell'ambito di una popolazione pediatrica. I batteri del genere Legionella restano una delle cause più frequenti di polmonite, anche se l'incidenza è certamente maggiore nell'età adulta rispetto a quell'infantile. Recentemente la possibilità di utilizzare metodi molecolari per la diagnosi di polmonite di Legionella ha portato ad un aumento del numero di casi diagnosticati, data la maggiore sensibilità dei test NAATS rispetto all'isolamento colturale. L'identificazione del DNA delle legionelle è possibile su campioni respiratori profondi e su urine. Sempre su campioni urinari è possibile eseguire test immunocromatografici per la messa in evidenza dell'antigene di Legionella in modo molto specifico e sensibile e, vista la minore complessità ed il minor costo del test immunocromatografico rispetto alle tecniche NAATs, la ricerca dell'antigene urinario di Legionella sta assumendo un ruolo sempre più rilevante nella diagnosi di polmonite con sospetta eziologia da legionelle (Murdoch, APMIS 112: 713 - 727; 2004).
Virus patogeni: virus respiratorio sinciziale, adenovirus, virus influenzali e parainfluenzali.
Dati epidemiologici dimostrano che l'eziologia virale è causa di circa il 70% delle polmoniti di comunità acquisite in età pediatrica ed in particolare i virus maggiormente implicati sono il virus respiratorio sinciziale (RSV) i virus influenzali A e B, i virus parainfluenzali e alcuni sierotipi di adenovirus. Con la sola eccezione degli adenovirus che hanno un genoma costituito da DNA, si tratta di RNA virus appartenenti alle famiglie dei paramyxovirus (RSV e parainfluenzali) ed orthomyxovirus (influenza A e B). Oltre alla polmonite questi virus sono causa di altre patologie respiratorie, in primo luogo la bronchiolite causata da RSV. Tecniche NAATs sono state sviluppate per ciascuno dei virus sopra elencati ed, invariabilmente i metodi PCR si sono dimostrati in grado di identificare un maggior numero di pazienti positivi rispetto alle tecniche di identificazione diretta (isolamento o immunofluorescenza) degli antigeni virali, verosimilmente grazie alla maggiore sensibilità diagnostica. Uno studio recente condotto su pazienti pediatrici ospedalizzati con diagnosi di polmonite, è stato dimostrato che l'applicazione di una metodica PCR aumenta in modo significativo il numero di casi identificati sia rispetto a quanto messo in evidenza con la tecnica di immunofluorescenza, sia rispetto al numero di casi diagnosticati con isolamento colturale. Sempre in questo studio è riportato che ben il 21% dei casi sono stati identificati unicamente mediante impiego della PCR. Dato che il quadro clinico e radiologico delle infezioni respiratorie virali risulta praticamente identico pur avendo eziologie differenti, sono stati condotti molti studi per la messa a punto di tests PCR multiplex, sia in formato "classico" che con metodologia real time, che consentano di studiare simultaneamente le differenti possibilità eziologiche (Murdoch, APMIS 112: 713 - 727; 2004).
Lo studio di seguito descritto è stato condotto al fine di valutare la performance diagnostica di un nuovo metodo basato sulla tecnologia Real Time PCR eseguito su piattaforma Light Cycler (Roche Diagnostic Systems) per la diagnosi di infezione da C.pneumoniae, M.pneumoniae, adenovirus e RSV in pazienti di età pediatrica. Il metodo impiegato è basato su primers specie-specifici e sulla tecnologia delle sonde FRET (fluorescence resonance energy transfer). I risultati sono stati comparati con quelli ottenuti con un metodo commerciale basato sulla metodica nested PCR per C.pneumoniae e M.pneumoniae. I risultati ottenuti per la determinazione della presenza di adenovirus sono stati comparati con quelli di una metodica commerciale "nested PCR" mentre il metodo di riferimento per RSV è stata una metodica di immunofluorescenza diretta. Popolazione studiata. Sono stati studiati 81 pazienti d'età pediatrica (1-10 anni) ricoverati per sintomi attribuibili ad infezione acuta respiratoria presso la Clinica Pediatrica dell'Ospedale di Cesena e da cui sono stati ottenuti 81 campioni di secreto faringeo. In aggiunta sono stati studiati 100 pazienti ricoverati presso la Clinica Pediatrica dell'Università di Bologna con diagnosi di sindrome respiratoria acuta a carattere bronchiolitico per la identificazione di RSV ed adenovirus. Ogni paziente ha avuto conferma radiologica del sospetto d'infezione acuta delle alte/basse vie respiratorie. I campioni faringei sono stati prelevati con tamponi in Dacron, posti in tubi contenenti terreno 2SP e congelati a -20°C fino all'esecuzione dei test diagnostici. I dettagli della popolazione studiata sono indicati nella tabella 1.

Valutazione di sensibilità e specificità del metodo. Per valutare la specificità del metodo sono stati utilizzati i seguenti microrganismi (cresciuti in vitro in adatti terreni di coltura) M. pneumoniae (ATCC 15377), M. hominis, M. fermentas, M. genitalium, Ureaplasma urealyticum Legionella pneumophila, Haemophilus influenzae, Streptococcus pneumoniae, Moraxella catarrhalis, Klebsiella pneumoniae. E' stata inoltre valutata la reattività di 4 isolati clinici di C. pneumoniae, 11 ceppi di C. psittaci (il ceppo 6BC e isolati di origine aviare), un ceppo di sierotipo D di C.trachomatis fatti crescere secondo il metodo in precedenza descritto ( ). Per valutare la sensibilità del metodo è stata allestita una diluizione seriale in base 10 di una sospensione batterica di M. pneumoniae fatto crescere in terreno SP-4 a 37°C per un periodo di due mesi al fine di valutare la carica batterica presente: il titolo della coltura è stato espresso in termini di Unità cambianti il colore (color-changing units CCU) per millilitro, essendo una CCU corrispondente a d un numero di cellule batteriche variabile da 10 a 100. C. pneumoniae (un recente isolato ottenuto presso il nostro laboratorio dal secreto faringeo di un soggetto affetto da faringite acuta) è stata fatta crescere in colture di cellule LLC-Mk2, purificata secondo il metodo descritto in precedenza ( ) e l'infettività della sospensione di corpi elementari (elementary bodies - EB) valutata mediante titolazione in colture cellulari e conta della unità formanti inclusione (ICU - Inclusion Forming Unit) mediante osservazione microscopica dei monostrati cellulari dopo 96 ore dall'infezione secondo quanto riportato in precedenza (Sambri et al., Vaccine 12: 1131-1137; 2004). La concentrazione della sospensione di EB è stata quindi aggiustata a 107 EB/ml. Da un volume di 50 ml di questa sospensione (quindi un numero totale di 5x108 EB) è stata eseguita l'estrazione del DNA totale secondo il metodo descritto di seguito e l'acido nucleico estratto è stato impiegato per la determinazione della sensibilità del metodo. La valutazione della sensibilità del test RT PCR per l'identificazione di adenovirus e RSV è stata eseguita valutando sospensioni cellulari infettate con un ceppi virali di collezione isolati brochiolite nel nostro laboratorio da pazienti affetti da patologia respiratoria acuta e la cui infettività è stata valutata come riportato da Falsey et al. (Journal of Clinical Microbiology, 41: 4160-4165; 2003). Al fine di saggiare la specificità del metodo è stata valutata la possibilità di risultati positivi per reattività crociata con viurus influenzali A e B e virus parainfluenzali. Tutti questi materiali sono stati preparati dal nostro laboratorio secondo le tecniche viro-logiche standard.
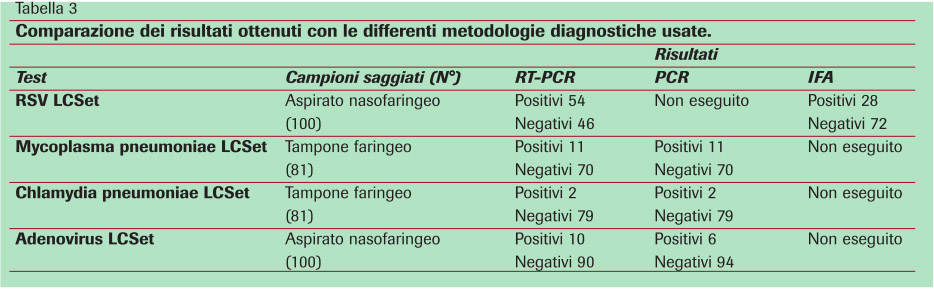
Risultati e discussione. I risultati ottenuti con le differenti metodiche impiegate sono riassunti nella Tabella 3. In dettaglio, la diagnosi di infezione da adenovirus è stata ottenuta nel 6% dei casi con il metodo nested PCR e nel 10% con il sistema RT-PCR, mentre RSV è stato identificato in 28 pazienti con la tecnica di immunofluorescenza diretta e in 54 con la metodica RT-PCR. Per quanto riguarda la diagnosi di infezione da batteri atipici (C.pneumoniae e M.pneumoniae) le percentuali di positività identificate nella popolazione studiata si sono dimostrate identiche per ciascuna delle metodiche utilizzate: 13,6% (11/81) per M.pneumoniae e 2,5% (2/81) per C.pneumoniae. La valutazione della specificità dei metodi RTPCR ha dimostrato che si tratta di tecniche ad alta specificità, poiché in nessun caso sono stati identificati risultati positivi in caso d'applicazione delle metodiche a campioni contenenti microrganismi differenti da quelli target. Inoltre la valutazione della sensibilità analitica di queste metodiche ha consentito di dimostrare che i metodi RTPCR studiati sono altamente sensibili, in quanto il numero minimo di microrganismi identificabili è di 50 CCU ml-1 per M. pneumoniae (corrispondenti a circa 2-20 organismi per reazione) e di 0.001 IFU di C. pneumoniae (corrispondenti a meno di 10 copie di genoma per reazione). Anche nel caso di adenovirus e RSV la sensibilità analitica è stata stabilita nel medesimo range (fra 10 e 30 copie di genoma target per reazione). Al fine di valicare il metodo di estrazione degli acidi nucleici e di monitorare la possibilità di risultati falsamente negativi dovuti alla presenza di inibitori della reazione di PCR, è stato aggiunto ad ogni test, a partire dalla prima fase del procedimento di estrazione, un controllo esterno costituito da plasmide lambda ed è stata eseguita un'amplificazione separata al fine di verificarne la corretta amplificazione: in nessun caso si sono avuti problemi connessi alla presenza di fenomeni inibitori la reazione enzimatica. Il quadro clinico dei piccoli pazienti affetti da infezioni respiratorie virali o da agenti batterici atipici non consente di porre con certezza la diagnosi eziologia e di stabilire la conseguente, eventualmente necessaria terapia antibiotica specifica nel caso di infezione batterica. Allo stesso modo i parametri di laboratorio, come la conta delle cellule bianche del sangue periferico,
o anche lo stesso quadro radiologico non sono sufficientemente informativi ai fini di stabilire con certezza l'eziologia della patologia respiratoria. Sulla base di tali considerazioni di ordine generale e sull'esperienza di questo studio, nel quale la metodologia RT-PCR ha dimostrato specificità elevatissima associata a sensibilità maggiore od eguale alle tecniche immunologiche o molecolari di più vecchia concezione, si può concludere che l'applicazione di questi metodi RT-PCR alla diagnostica delle infezioni respiratorie in età pediatrica presenta svariati vantaggi, sotto delineati analiticamente.
1. La rapidità di esecuzione della metodica, associata alle elevate sensibilità e specificità, consente di porre la diagnosi eziologia in tempi molto ravvicinati rispetto all'ingresso del piccolo paziente in ambito ospedaliero.
2. La rapidità di diagnosi consente di porre in atto in tempi ristretti la terapia antibiotica e permette quindi una potenziale riduzione dei tempi di permanenza in ospedale in attesa di diagnosi eziologia.
4. Dal punto di vista più strettamente laboratoristico, la metodologia RT-PCR si è inoltre confermata semplice da eseguire. Inoltre, grazie alla lettura automatica dei risultati ottenuti e all'interpretazione degli stessi mediante il software dedicato si è dimostrata di grande applicabilità pratica alla diagnosi di laboratorio delle infezioni respiratorie da patogeni atipici, anche in laboratori che debbano affrontare giornalmente cospicui numeri di richieste di indagini dagnostiche.